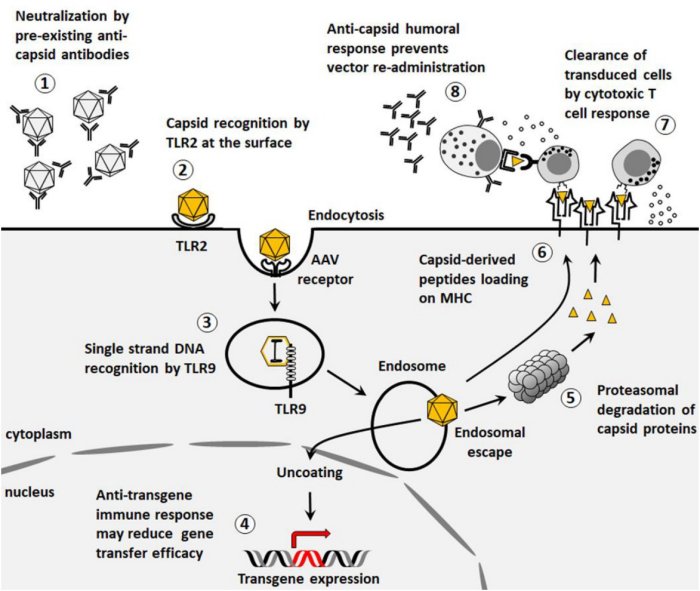
Christian Mueller et son équipe chez Sanofi ont développé ce modèle qui évalue la « forme physique » des capsides AAV2 en s’appuyant sur la séquence d’acides aminés de leur monomère de capside.
En intégrant des techniques de machine learning classiques avec un modèle de langue protéique (PLM), ils ont remarquablement atteint une précision de prédiction élevée, comme en témoigne une corrélation de Pearson de 0,818.
Cette avancée est particulièrement significative car elle a également démontré une forte robustesse lors des tests sur des datasets complètement indépendants, y compris pour les capsides avec de multiples mutations.L’utilisation de l’intelligence artificielle pour améliorer le design des capsides représente un tournant potentiel pour rendre les thérapies géniques plus accessibles et moins onéreuses.
Traditionnellement, le développement des capsides nécessitait des approches empiriques coûteuses comme l’évolution dirigée ou la conception rationnelle.
Le modèle présenté par l’équipe de Sanofi offre une alternative moins coûteuse et peut potentiellement accélérer le développement de thérapies géniques plus efficaces.
Par ailleurs, selon l’étude de Wu et al.de la Massachusetts Chan Medical School, cette méthode pourrait se généraliser à d’autres applications, promettant ainsi un impact plus large sur le domaine médical.
Cette recherche ne se contente pas de pousser les frontières scientifiques, elle ouvre également la voie à des traitements plus abordables pour les patients, marquant un progrès significatif dans la lutte contre de nombreuses maladies génétiques.